mimics新手教程.docx
《mimics新手教程.docx》由会员分享,可在线阅读,更多相关《mimics新手教程.docx(10页珍藏版)》请在冰点文库上搜索。
mimics新手教程
mimics10.01教程--入门级
(二)------import导入
2011-05-110:
24
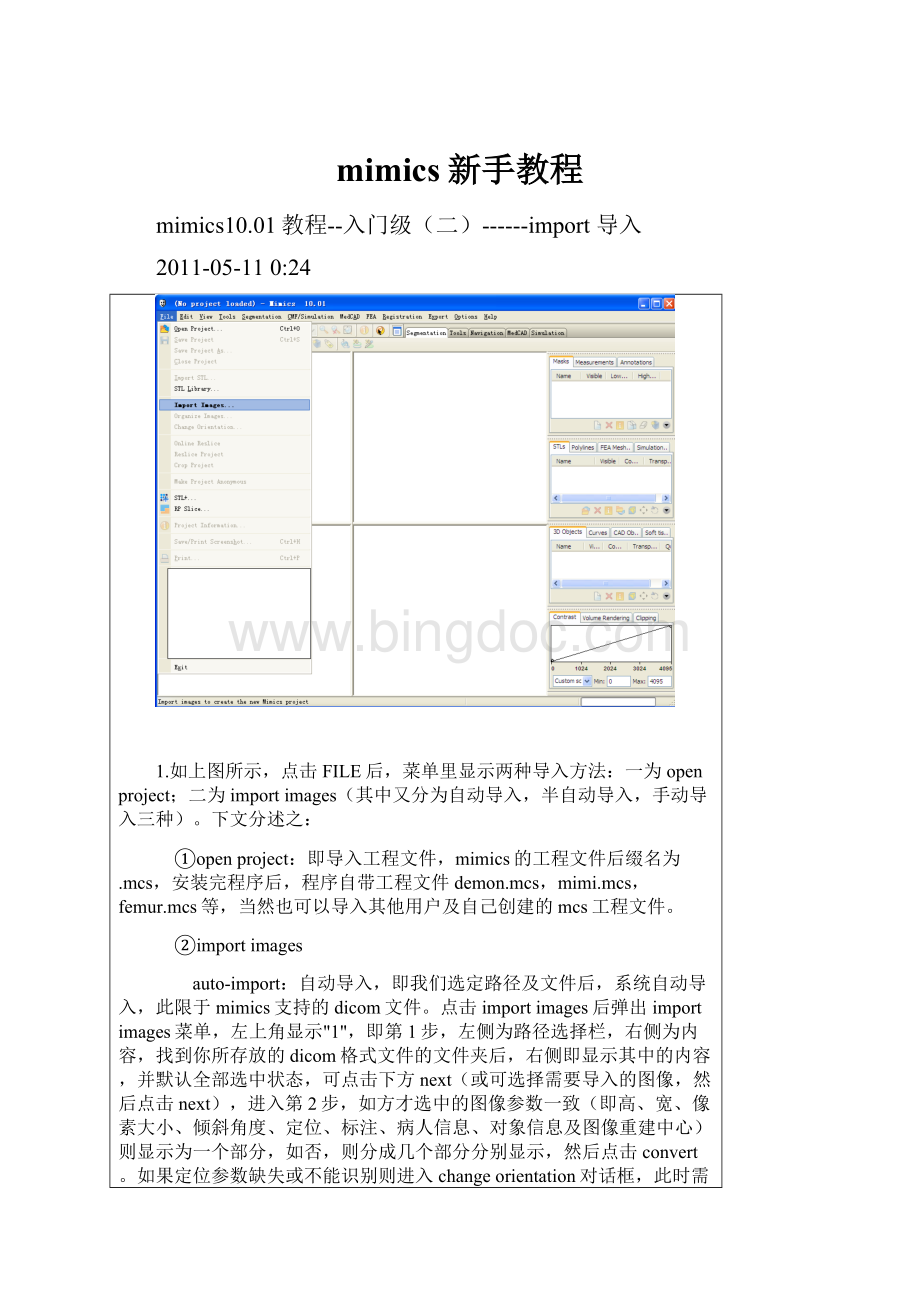
1.如上图所示,点击FILE后,菜单里显示两种导入方法:
一为openproject;二为importimages(其中又分为自动导入,半自动导入,手动导入三种)。
下文分述之:
①openproject:
即导入工程文件,mimics的工程文件后缀名为.mcs,安装完程序后,程序自带工程文件demon.mcs,mimi.mcs,femur.mcs等,当然也可以导入其他用户及自己创建的mcs工程文件。
②importimages
auto-import:
自动导入,即我们选定路径及文件后,系统自动导入,此限于mimics支持的dicom文件。
点击importimages后弹出importimages菜单,左上角显示"1",即第1步,左侧为路径选择栏,右侧为内容,找到你所存放的dicom格式文件的文件夹后,右侧即显示其中的内容,并默认全部选中状态,可点击下方next(或可选择需要导入的图像,然后点击next),进入第2步,如方才选中的图像参数一致(即高、宽、像素大小、倾斜角度、定位、标注、病人信息、对象信息及图像重建中心)则显示为一个部分,如否,则分成几个部分分别显示,然后点击convert。
如果定位参数缺失或不能识别则进入changeorientation对话框,此时需手动设置图像的方向,移动鼠标至图像中标为"X”的部位,右键单击,选择top或bottom等。
设置完成,点击OK。
Semi-automaticimport:
半自动导入,当导入的文件格式为BMP或TIFF时,会弹出BMP/TIFFimport对话框,以设置部分参数,见下图:
一般手动更改红色箭头所标示的SliceDistance,PixelSize数值,然后点击OK即可,,如导入错误,则说明其余数值有变动,应向CT扫描人员索取变动信息并填入,余同前。
可预先preview。
manualimport:
手动导入,当在importimages菜单的第一步时,将对话框下方的manualimport选中并点击next,
当导入的文件为多个时,则弹出下图对话框,将其中内容填写后点击ok即可。
可预先preview。
说明:
FileheaderSize 头文件大小,mimics可自动计算。
如需手动填写,可有公式计算,此处从略。
ExamInformation中:
1)Slices:
医用扫描一般为横断面成像,默认选择Axial(轴视图)。
ImageInformation中:
2)Size:
是图像的大小。
3)SliceDistance:
层距。
4)PixelSize:
像素的大小,可从像素间距得出。
如像素间距为0.8398x0.8398,则此处填0.8398.
PixelProperties中:
5)Type:
选择Signedshort或者Unsignedshort。
当导入的文件为一个文件时,比如一些裸数据文件,如(http:
//www.volvis.org/网站,然后点击DownloadNewDataset后可下载)stent16.raw文件,弹出如下图示:
说明:
FileStructure中:
1)FileHeaderSize:
指图像文件头部的大小。
裸数据文件没有头部信息只包含像素,所以填0。
2)NumberofImagesperFile:
是指裸数据文件中一共包含的图像数。
mimics10.01教程--help之中文翻译--中文帮助--mimicstutorial-Mimi--Windowing
2011-05-1112:
09
Windowing
视窗调整
Firstofall,wehavetoadjustthecontrastoftheimagesdisplayedinthedifferentviews.Contrastenhancementisaverygoodtoolforselectingpartswithdifferentintensities,e.g.bonevs.braintumor.Thisactioncanbeperformedatanytime.
首先,我们需要在不同的视窗中调整图像的对比度,对于选择不同密度的选区来说,对比度增强是一个很好的工具。
举例来说,如骨与脑肿瘤。
这个功能在任何时候均可使用。
YoucanchangethecontrastinthecorrespondingtaboftheProjectmanagement.Thecontrasttabshowsthehistogramoftheprojectwithalinerepresentingthe“window”.ThegrayvaluesorHounsfieldunitsbelowthestartpointofthelinewillbedisplayedinblack.Allgrayvaluesabovetheendpointofthelinewillbedisplayedinwhite.Thegrayvaluesinbetweenthewindowwillbemappedonashadeofgray.
你可以在相关工具栏里更改工程管理的对比度。
对比度工具栏将工程以直方图方式显示,并以一条线代表了视窗的调整。
比这条线起始点低的灰度值或亨氏单位被以黑色显示,反之,比此线结束点高的以白色显示。
居中的灰度值以灰色的明暗显示。
Youcanchangethewindowsizebyclickingyourleftmouseononeofthepointsanddraggingittoitsnewlocation.Tomovethewindowselectthelineanddragittoitsnewposition.
你可以左键点击起始点或结束点中的一个并将其拖动到新的位置来做视窗的调整。
也可点击代表视窗的那条线并拖动至新位置来调整。
Youcanalsochooseoneofthepredefined“windows”byselectingtheappropriatescalefromthemenuonthebottomofthetab.
你也可以通过挑选此栏底部的适当的刻度值,来选择视窗调整的预设值。
Thefollowingstepswilldescribethenecessaryactionstoachieveanicesegmentationmask.Asegmentationmaskisacollectionofpixelsofinterestthatconstituteanobjectyouwishtoworkon.Onecancreateseveral-dependentorindependent-masks,eachdisplayedwiththeirownidentifyingcolor.Usuallyseveralmaskswillbeneededtoobtainafinalsegmentationobjectthatcontainstheinformationthatisneeded.
下面的步骤将描述必要的命令,以便于更好地进行蒙罩分割。
分割后的蒙罩即为你感兴趣的像素集,它包含着你希望处理的对象。
你可以建立几个独立的或非独立的蒙罩,每个都可以以可辨识的颜色来显示。
通常蒙罩应为包含所需信息的分割图像。
mimics10.01教程--入门级(三)------Windowing视窗调整
2011-05-1112:
58
mimics中所提到的windowing,指的是调整视窗图像的对比度,使感兴趣的组织突出显示,以便于观察。
调节栏在右下角的contrast,以直方图的方式,其中又两个关键点,分别为起始点和结束点,调整两点的位置,可以观察到视窗图中的变化。
可以看到:
低于左边点密度的组织呈黑色,左边点的位置调向极右,则连骨组织也成黑色;高于右边点密度的组织呈白色,右边点的位置调向极左,则体内空气亦成白色;向上调节两点,图像整体变白,反之则变黑;两点上下距离越大,对比越大,反之对比越小,图像内容无法区分。
简单来说:
点向右,则黑多白少,点向左,则黑少白多;点向上,则白,点向下,则黑;点距越大,对比越大。
mimics10.01教程--入门级(四)------Thresholding阈值分割
2011-05-1113:
23
如本例以mimi.mcs工程文件为例,提取颅骨为目的。
1.点击如上图所示按钮,即Thresholding 阈值分割按钮,并在轴视图中选取要提取的组织(分别在感兴趣的组织两侧左键单击即可,软件会自动生成一条线段横跨目标组织,如图示1)。
2.弹出profilelines对话框,点击startthresholding(如图示2),mimics会自动分析并在左上方图示中表现出来。
3.并弹出对话框thresholding(如图示3)
4点击apply按钮,并关闭对话框,可以看到,在3个视图里(轴视图、侧视图、前视图)颅骨组织被提取出来了,并以默认的颜色以示区分,同时在右上角Marks栏里将此次结果定义为蒙罩,起名为Green,并visible状态显示为可见,及最低阈值1250,最高阈值3810.
mimics10.01教程--入门级(五)------Regiongrowing区域增长
2011-05-1114:
04
滑动每个视窗右侧的滑块观察各个层面的图像,可以发现,除外颅骨,还有一些其他的区域被选了进来,我们称之为噪点及误选区,此时将用的Regiongrowing 区域增长功能。
1.点击mimics界面左上角区域增长按钮,如下图图示1。
2.弹出RegionGrowing对话框,目标区域默认为newmask(我这里已经做完这步,所以显示yellow)。
3.在任意视图中的绿色区域内点击要选取的组织,如在轴视图中点击已被绿色遮盖的颅骨横断面(其实质是在已选取的组织中提取连续的组织,不连续的组织将被摒弃),则可以发现,颅骨被选中而显示为新蒙罩的黄色,其余噪点及误选区如皮肤等因为和颅骨不相连,故未被选取,如此,颅骨就被“提取”出来了,成为一个新的蒙罩。
mimics10.01教程--入门级(六)------Calculatinga3D3D重建
2011-05-1317:
16
1.如上图,在选中要3D重建的蒙罩(即对象,本例为yellow)的状态下,点击其右下角的Calculate3D frommask按钮(即图中1处)。
;
2.弹出Calculate3D 对话框(见图中2处),默认选择optimal(最优的),点击calculate;
3.稍等片刻,在3D视图中便可见到重建后的3D模型(如没有,可点击两次图中4处),此为面数据。
在右侧的3D信息栏中可以看到自动命名的3D对象,处可视状态。
4.在3D视图中,可以通过右键悬于视图中时,按住不放调整角度,鼠标右键+Ctrl可放大或缩小目标对象,鼠标左键+Shift可平移目标图像。
mimics10.01教程--入门级(七)------Remeshingthe3D3D面网格化
2011-05-1323:
58